Frontiers in Genetics
Einführung
Small Open Reading Frames (kleine ORFs/sORFs/smORFs) sind wichtige Quellen für mutmaßliche Peptide, die früher als nicht-funktionale oder Junk-DNA abgetan wurden, wie frühe Methoden der Genvorhersage zeigten. Tatsächlich sind smORFs von <100 Codons mögliche kodierende Sequenzen, aber ausreichend klein, um sehr häufig und zufällig in Genomen aufzutreten; daher gleicht der Nachweis ihres kodierenden Potenzials und die funktionelle Bewertung einem Spaziergang im Dunkeln. Darüber hinaus wurden in letzter Zeit zwar Dutzende von smORF-Peptiden als essentielle Akteure in biologischen Prozessen beschrieben, aber viele von ihnen werden als potenzielle nicht-funktionelle Produkte von Junk-DNA unter durchdringender Translation beschrieben, was zu der Frage führt: Aus welcher Perspektive wird dieser Mangel an Funktion bewertet? In diesem Zusammenhang wurde vor kurzem vorgeschlagen, dass nicht-funktionale smORF-Peptide eine wichtige Rolle bei der de novo Protein kodierenden Gengeburt spielen könnten, aber der evolutionäre Mechanismus ist noch unklar. Somit bleibt die Rolle der durchdringenden Translation von smORFs in der molekularen Evolution rätselhaft. Hier stellen wir interessante Fragen zur Diskussion und weiteren Untersuchung über die Perspektive von nicht-funktionalen smORF-Peptiden als unterschätzte Hotspots der molekularen Evolution in Eukaryoten.
Small Open Reading Frames: Ein Unterthema in der Diskussion der Junk-DNA-Funktion
Im Hinblick auf die Evolution der molekularen Funktion akkumuliert ein Teil der DNA-Elemente Mutationen durch genetische Drift; die Evolution dieser Elemente ist also nicht-adaptiv und neutral (Ohta, 2002). In einigen Fällen ist die Menge der neutral evolvierenden Elemente in der Junk-DNA analog zu den Elementen auf einer Speisekarte, die der natürlichen Selektion zur Verfügung stehen (Knibbe et al., 2007; Faulkner und Carninci, 2009; Lynch et al., 2011). Interessanterweise wurde vom ENCODE-Konsortium (Encyclopedia of DNA Elements) berichtet, dass der größte Teil der menschlichen Junk-DNA irgendeine Art von biochemischer Aktivität aufweist (ENCODE Project Consortium, 2012), jedoch ohne adaptive Relevanz und Selektionsdruck (Doolittle, 2013; Graur et al., 2013). Wichtig ist, dass Junk-DNA 75-90 % des menschlichen Genoms ausmacht (Graur, 2017).
Ein Teil des Junk-DNA-Menüs besteht aus neutral evolvierenden smORF-Peptiden. So werden Tausende von nicht-kodierenden RNAs durch die umfangreiche Transkriptionsabdeckung auf Junk-DNA erzeugt (ENCODE Project Consortium, 2007). Es gibt immer mehr Hinweise darauf, dass Tausende von smORFs in Transkripten, die als nicht-kodierend annotiert sind, oder in den untranslatierten Regionen (UTR) von mRNAs eine weitreichende Translation erfahren (z. B. Aspden et al., 2014; Ingolia et al., 2014). Interessanterweise wurde berichtet, dass nicht-kodierende RNAs und ORFs ohne Homologe Kandidaten für die de novo-Evolution von proteinkodierenden Genen sind (Tautz und Domazet-Lošo, 2011). Darüber hinaus wurde kürzlich vorgeschlagen, dass neutral evolvierende smORF-Peptide eine wichtige Rolle in diesem Prozess spielen könnten (Ruiz-Orera et al., 2018), aber der evolutionäre Mechanismus muss noch bestimmt werden (Ruiz-Orera et al., 2018; Singh und Wurtele, 2020). In diesem Zusammenhang stehen zwei zuvor vorgeschlagene Konzepte zur Diskussion der Evolution molekularer Funktionen im Mittelpunkt der Junk-DNA-Debatte: „kausale Rollen“ und „selektierte Effekte“ (Doolittle und Brunet, 2017), die hier im Kontext von smORFs und der Geburt von proteinkodierenden Genen diskutiert werden sollen.
Die „kausale Rolle“ beschreibt die Aktivität, die ein neutral evolvierendes Element zufällig ausübt. Zum Beispiel kann eine hypothetische genomische Sequenz, die durch eine zufällige Nukleotidmutation erzeugt wird und einer TATA-Box ähnelt, von Transkriptionsfaktoren erkannt und gebunden werden, löst aber keine Gentranskription aus (Griffiths, 2009; Graur et al., 2013). Mit anderen Worten, „kausale Rollen“ sind nicht-adaptive Phänotypen, ihr Auftreten ist zufällig, und sie neigen dazu, im Laufe der Evolution schnell zu verschwinden. Auf der anderen Seite beschreiben „selektierte Effekte“ den Erwerb von adaptiven Phänotypen aufgrund natürlicher Selektion (Graur et al., 2013), wie z. B. kanonische TATA-Boxen oder ORFs, die in wichtige Proteine übersetzt werden. Mit anderen Worten: „Selektierte Effekte“ sind funktionell relevant für Zellen.
Während die natürliche Selektion die adaptive Evolution antreibt (selektierte Effekte), ist es weithin akzeptiert, dass die genetische Drift die Junk-DNA-Evolution antreibt, ebenso wie die synonymen Modifikationen in kodierenden DNA-Sequenzen (CDS) und Mutationen in UTRs von mRNAs (Ridley, 2004).
Diskussion
Anhand der oben genannten Beweise und Konzepte diskutieren wir hier einen möglichen eukaryotischen Mechanismus, durch den neutral evolvierende smORFs die Proteomevolution vorantreiben und die evolutionäre Bedeutung von smORFs.
Erstens geht ein Teil der von neutral evolvierenden smORF-Peptiden ausgeübten Rollen unter Umweltdruck möglicherweise von „kausalen Rollen“ zu „ausgewählten Effekten“ über, wodurch ihre neutralen Phänotypen der natürlichen Selektion ausgesetzt werden und die Evolution neuer kodierender Gene ausgelöst wird. Wenn also neutrale smORF-Peptide selektiert werden, sind sie nicht mehr neutral (Ruiz-Orera et al., 2018). Mit anderen Worten, neutrale smORF-Peptide können besondere Vorspeisen auf dem Junk-DNA-Menü sein, die der natürlichen Selektion zur Verfügung stehen (Abbildung 1A).
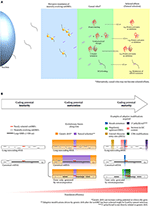
Abbildung 1. Phänotypselektion und Reifung des kodierenden Potenzials von smORF-Transkripten. (A) Übergang von smORF-Peptiden von „kausalen Rollen“ zu „ausgewählten Effekten“ nach pervasiven Translationsereignissen. Pervasive Translation von neutral evolvierenden smORFs treibt möglicherweise die Proteomevolution voran, indem neutrale Phänotypen unter Umweltdruck der natürlichen Selektion ausgesetzt werden. (B) Schema für die Reifung des Kodierungspotenzials, ein hypothetischer Mechanismus, der die Translationseffizienz einer mRNA erhöht, nachdem ein smORF in einem Transkript mit suboptimalen Kodierungseigenschaften selektiert wurde (selektierter Effekt). Links: Unreife des Kodierungspotenzials; in der Mitte: Reifung des Kodierungspotenzials; rechts: Reifung des Kodierungspotenzials. Während der Phase der Unreife des Kodierungspotenzials werden neu selektierte smORFs in Transkripten mit suboptimalen Kodierungseigenschaften beobachtet, entweder in langen nicht-kodierenden RNAs oder als alternative smORFs in kanonischen mRNAs. Obwohl kanonische mRNAs optimale Kodierungsmerkmale aufweisen, werden alternative smORFs in der Regel sekundär oder pervasiv translatiert; daher können sich einige alternative smORFs in suboptimalen Kodierungsregionen befinden. Während der Reifungsphase des kodierenden Potenzials können natürliche Selektion und genetische Drift in verschiedenen Teilen eines Transkripts wirken. Während die natürliche Selektion durch Fixierung der selektierten Teile wirkt, wirkt die genetische Drift durch Veränderung der nicht-kodierenden Teile eines Transkripts, wie es die nahezu neutrale Theorie postuliert (Ohta, 2002). Die natürliche Selektion fördert fein abgestimmte Anpassungen an die selektierten Phänotypen, wie synonyme Mutationen und CDS-Modifikationen. Genetische Drift kann adaptive Mutationen in einem Transkript etablieren, indem Sequenzen evolviert werden, die potentiell die smORF-Translation erhöhen, wie z. B. der Kozak-Konsensus, regulatorische Upstream-ORFs, interne Ribosomeneintrittsstellen (IRES) und Erhöhungen des GC-Gehalts. Zusätzlich können andere adaptive Modifikationen, die nicht direkt mit Sequenzmutationen in Transkripten zusammenhängen, die smORF-Expression erhöhen, wie z. B. die 5′-Kappe, der 3′-Poly(A)-Schwanz, cis-regulatorische Elemente im Genom und, im Fall von alternativen smORFs, die Erzeugung unabhängiger Geneinheiten durch Retrotransposition. Wichtig ist, dass der Erwerb optimaler kodierender Eigenschaften begünstigt werden könnte, nachdem der smORF selektiert wurde, weil durch genetische Drift getriebene Modifikationen durch natürliche Selektion fixiert werden könnten, wenn sie die Translationseffizienz des neu selektierten smORFs verbessern. Bevor der smORF selektiert wurde, könnten eventuell erworbene optimale Kodierungseigenschaften während der Evolution durch genetische Drift schnell verschwinden, ohne dass eine Fixierung erfolgt. Alternativ können Mutationen, die durch genetische Drift entstanden sind, das Gen zum Schweigen bringen. Schließlich erreichen smORFs die Reifephase des Kodierungspotenzials, wenn optimale Kodierungsmerkmale erworben werden und die Translationseffizienz steigt. Folglich wird die Translationsrate von smORF-Peptiden nach Abschluss des beschriebenen Prozesses stark erhöht, was zur Etablierung von molekularen Innovationen und zur Geburt von proteinkodierenden Genen beiträgt.
Wenn smORFs selektiert werden, haben sie aufgrund ihrer nicht-kodierenden Transkriptmerkmale, wie z. B. geringe Translationsrate, fehlende 3′-terminale Prozessierung und andere suboptimale kodierende Merkmale, wahrscheinlich eine geringe adaptive Relevanz (nicht-kodierende RNA-Merkmale werden in Quinn und Chang, 2016, besprochen). Diese Hypothese basiert auf der Tatsache, dass Hunderte von smORFs als hoch konserviert beschrieben werden, aber eine niedrige Expression und Translationseffizienz aufweisen und in Transkripten mit nicht-kodierenden Eigenschaften beobachtet werden (Cabili et al., 2011; Aspden et al., 2014; Bazzini et al., 2014). Die nahezu neutrale Theorie (Ohta, 2002) legt jedoch nahe, dass nicht-kodierende Teile fester smORF-Transkripte durch zufällige genetische Drift modifiziert werden, was in einigen Fällen zu kleinen vorteilhaften (oder nachteiligen) adaptiven Effekten im Laufe der Evolution führt; daher schlagen wir vor, dass diese Modifikationen an einem bestimmten Punkt das kodierende Potenzial von smORF-Transkripten verfeinern und erhöhen und folglich die adaptive Relevanz ihrer Peptide verbessern, wie dies bei einer großen Anzahl wichtiger smORF-Peptide, die kürzlich entdeckt wurden, zu sehen ist (z. B., Magny et al., 2013; Anderson et al., 2015; Lauressergues et al., 2015; Nelson et al., 2016; Pengpeng et al., 2017; Kim et al., 2018; Polycarpou-Schwarz et al., 2018; Chugunova et al., 2019; Tobias-Santos et al., 2019; Pang et al., 2020; Vassallo et al., 2020). Wichtig ist, dass der Erwerb mehrerer optimaler Kodierungsmerkmale begünstigt werden könnte, nachdem der smORF selektiert wurde, da durch genetische Drift getriebene Modifikationen durch natürliche Selektion fixiert werden könnten, wenn sie die Translationseffizienz des neu selektierten smORFs verbessern. Bevor der smORF selektiert wurde, könnten eventuelle optimale kodierende Merkmale, die in der Nukleotidsequenz erworben wurden, während der genetischen Drift-Evolution ohne Fixierung schnell verschwinden. Alternativ können Nukleotidveränderungen das kodierende Potenzial negativ beeinflussen und ein Gen zum Schweigen bringen. Zu den optimalen Kodierungsmerkmalen gehören die strukturelle Stabilisierung, das Auftreten von Kozak-Konsensus, interne Ribosomeneintrittsstellen (IRES), die Abdeckung durch Enhancer und in einigen Fällen die Verlängerung von kodierenden smORFs, um die CDSs (kodierende DNA-Sequenzen) zu vergrößern (Abbildung 1B). Kürzlich schlugen Couso und Patraquim (2017) vor, dass zumindest ein Teil der funktionellen smORFs potenzielle de novo-Vorläufer großer CDSs sind, und zwar über ein Stopcodon-Mutationsmuster, das als „CDS-Elongation“ bezeichnet wird.
In Anbetracht der Annahme, dass die Evolution schrittweise abläuft, schlagen wir vor, den oben genannten Prozess als „Reifung des kodierenden Potenzials“ zu bezeichnen (Abbildung 1B). Zum Beispiel wird häufig über smORF-Translation in Transkripten mit Eigenschaften einer langen nicht-kodierenden RNA (lncRNA) berichtet (Crappé et al., 2013; Ingolia et al., 2014; Ji et al., 2015; Mackowiak et al., 2015; Li et al., 2018; Lu et al., 2019). Diese lncRNAs zeigen smORF-Konservierung in divergenten Spezies, was auf eine Fixierung durch natürliche Selektion hindeutet und auf kodierende Unreife hindeutet.
Ein weiterer möglicher Weg der kodierenden Gengenerierung erfolgt über alternative smORFs in UTRs oder überlappend mit dem Referenz-CDS kanonischer mRNAs. In diesem Szenario durchlaufen alternative smORFs eine durchdringende Translation oder der Akt der Translation selbst ist für cis-regulatorische Zwecke wichtig (Vanderperre et al., 2013; Wu et al., 2020). Wenn die „kausalen Rollen“, die von neutral evolvierenden smORF-Peptiden ausgeübt werden, zu „selektierten Effekten“ werden, würden die alternativen smORFs durch Retrotransposition unabhängige Geneinheiten erzeugen, oder sie würden als alternative smORFs in den ursprünglichen Transkripten fixiert werden (Abbildung 1B). Während Retrotranspositionsereignissen kann daher zumindest ein Teil der Transkripte, die auf der Basis von Pseudogenisierung untersucht wurden, tatsächlich die Reifung neuer kodierender Gene darstellen, wie ein Bericht nahelegt, dass Pseudogene in hochkonservierte smORF-Peptide übersetzt werden können (Ji et al., 2015).
smORFs könnten Sequenzreservoirs sein, die während der Evolution neuer phänotypischer Variationen, insbesondere während der Speziation, potenziell aktiviert werden. Wichtig ist, dass Speziationsereignisse mit der Evolution neuer molekularer Phänotypen und neuer Beziehungen mit der Umwelt verbunden sind (Bao et al., 2018). Daher verdient die Menge an Junk-DNA und lncRNAs in Zellen eine Untersuchung nicht nur als zufällige Anhäufung von Sequenzen und Translationsrauschen, sondern auch als Lagerstätte von Substraten, die die Evolution neuer kodierender Gene vorantreiben. Interessanterweise wurden Polyploidisierung oder Ganzgenom-Duplikationsereignisse (WGD) mit einem Anstieg des adaptiven Potenzials von Zellen und Organismen, die stressigen Bedingungen ausgesetzt sind, in Verbindung gebracht (Van De Peer et al., 2017). Leider haben Studien zu WGD bisher die Rolle und den Erhalt von smORFs während der Evolution vernachlässigt, was wahrscheinlich auf methodische Schwierigkeiten bei der smORF-Identifizierung zurückzuführen ist.
Die Sequenzierung mehrerer Genome auf der Grundlage vergleichender Ansätze hat jedoch kürzlich neue Wege für die smORF-Forschung eröffnet. Zum Beispiel haben kürzlich von unserer Gruppe durchgeführte evolutionäre Studien zu den smORFs im mille-pattes/tarsalless/polished rice (mlpt)-Gen, dem bekanntesten smORF-haltigen Gen bei Insekten (Savard et al, 2006; Kondo et al., 2007; Pueyo und Couso, 2008, 2011; Cao et al., 2017; Ray et al., 2019), zeigte, dass während der Evolution der Hemiptera ein neuer smORF mit ~80 Aminosäuren (smHemiptera) entstand (Tobias-Santos et al., 2019). Somit ist dieser smORF in der polyzistronischen mlpt mRNA seit über 250 Millionen Jahren in der Gruppe konserviert, und er ist in den Genomen anderer Insektenordnungen nicht vorhanden. Wir erwarten, dass neue vergleichende Analysen von Genomen in der Zukunft weitere Beispiele für ordnungsspezifische smORFs liefern werden, die ein unterschätztes Reservoir an neuen Genen und evolutionären Innovationen darstellen könnten.
Zusammenfassend lässt sich sagen, dass die Untersuchung von smORFs in den letzten 5 Jahren aufgrund der jüngsten Entdeckungen wichtiger smORF-Peptide erheblich zugenommen hat. Dementsprechend hat das Aufkommen der Ribosomen-Profilierung die Entdeckung vieler neutral evolvierender und potenziell nicht-funktionaler smORFs ermöglicht, die eine durchdringende Translation durchlaufen und deren Bedeutung noch zu bestimmen ist (Crappé et al., 2013; Aspden et al., 2014; Bazzini et al., 2014; Olexiouk et al., 2016). In diesem Zusammenhang stellt sich die faszinierende Frage: Warum sollten Zellen Energie für die Transkription und Translation von neutralen und nicht-funktionalen Elementen aufwenden? Wahrscheinlich gibt es mehr als eine Antwort; in Anbetracht der in diesem Artikel diskutierten Themen schlagen wir jedoch die folgende Perspektive vor: Was wäre, wenn die allgegenwärtige Translation von neutral evolvierenden smORF-Peptiden einen eleganten Mechanismus darstellt, um die Proteomevolution voranzutreiben, insbesondere während Speziationsereignissen? Wenn dem so ist, dann haben nicht-funktionale smORF-Peptide eine wichtige Funktion im evolutionären Sinne. Basierend auf dieser Diskussion schlagen wir vor, das Konzept der Funktionalität im Kontext von smORFs zu überarbeiten.
Beiträge der Autoren
DG-A und RN haben zu gleichen Teilen zum Schreiben dieses Manuskripts beigetragen. RN trug zur Beschaffung der finanziellen Mittel bei. Alle Autoren trugen zum Artikel bei und genehmigten die eingereichte Version.
Finanzierung
RN wurde von CNPq (307952/2017-7 und 431354/2016-2) und FAPERJ (E-26/210-150/2016, E-26/203.298/2016, E-26/202.605/2019, und E-26/211.169/2019) unterstützt. DG-A war Masterstudent der PPG-PRODBIO-UFRJ/Macaé (CAPES-Stipendium).
Interessenkonflikt
Die Autoren erklären, dass die Forschung in Abwesenheit von kommerziellen oder finanziellen Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Anderson, D. M., Anderson, K. M., Chang, C. L., Makarewich, C. A., Nelson, B. R., McAnally, J. R., et al. (2015). Ein Mikropeptid, kodiert von einer putativen langen nicht-kodierenden RNA, reguliert die Muskelleistung. Cell 160, 595-606. doi: 10.1016/j.cell.2015.01.009
PubMed Abstract | CrossRef Full Text | Google Scholar
Aspden, J. L., Eyre-Walker, Y. C., Phillips, R. J., Amin, U., Mumtaz, M. A. S., Brocard, M., et al. (2014). Extensive translation of small open reading frames revealed by poly-ribo-seq. eLife 3:e03528. doi: 10.7554/eLife.03528
PubMed Abstract | CrossRef Full Text | Google Scholar
Bao, R., Dia, S. E., Issa, H. A., Alhusein, D., and Friedrich, M. (2018). Vergleichende Belege für einen außergewöhnlichen Einfluss der Genduplikation auf die Entwicklungsevolution von Drosophila und den höheren Diptera. Front. Ecol. Evol. 6:63. doi: 10.3389/fevo.2018.00063
CrossRef Full Text | Google Scholar
Bazzini, A. A., Johnstone, T. G, Christiano, R., Mackowiak, S. D., Obermayer, B., Fleming, E. S., et al. (2014). Identifizierung von kleinen ORFs in Wirbeltieren mittels Ribosomen-Footprinting und evolutionärer Konservierung. EMBO J. 33, 981-993. doi: 10.1002/embj.201488411
PubMed Abstract | CrossRef Full Text | Google Scholar
Cabili, M., Trapnell, C., Goff, L., Koziol, M., Tazon-Vega, B., Regev, A., et al. (2011). Integrative Annotation von humanen großen intergenen nicht-kodierenden RNAs zeigt globale Eigenschaften und spezifische Subklassen. Genes Dev. 25, 1915-1927. doi: 10.1101/gad.17446611
PubMed Abstract | CrossRef Full Text | Google Scholar
Cao, G., Gong, Y., Hu, X., Zhu, M., Liang, Z. C., Huang, L., et al. (2017). Identification of tarsal-less peptides from the silkworm Bombyx mori. Appl. Microbiol. Biotechnol. 102, 1809-1822. doi: 10.1007/s00253-017-8708-4
PubMed Abstract | CrossRef Full Text | Google Scholar
Chugunova, A., Loseva, E., Mazin, P., Mitina, A., Navalayeu, T., Bilan, D., et al. (2019). LINC00116 kodiert für ein mitochondriales Peptid, das Atmung und Lipidstoffwechsel miteinander verbindet. Proc. Natl. Acad. Sci. U.S.A. 116, 4940-4945. doi: 10.1073/pnas.1809105116
PubMed Abstract | CrossRef Full Text | Google Scholar
Couso, J., and Patraquim, P. (2017). Klassifizierung und Funktion von kleinen offenen Leserahmen. Nat. Rev. Mol. Cell Biol. 18, 575-589. doi: 10.1038/nrm.2017.58
PubMed Abstract | CrossRef Full Text | Google Scholar
Crappé, J., Criekinge, W. V., Trooskens, G., Hayakawa, E., Luyten, W., Baggerman, G., et al. (2013). Combining in silico prediction and ribosome profiling in a genome-wide search for novel putatively coding sORFs. BMC Genomics 14:648. doi: 10.1186/1471-2164-14-648
PubMed Abstract | CrossRef Full Text | Google Scholar
Doolittle, W. F. (2013). Is junk DNA bunk? A critique of ENCODE. Proc. Natl. Acad. Sci. U.S.A. 110, 5294-5300. doi: 10.1073/pnas.1221376110
CrossRef Full Text | Google Scholar
Doolittle, W. F., and Brunet, T. D. P. (2017). On causal roles and selected effects: our genome is mostly junk. BMC Biol. 15:116. doi: 10.1186/s12915-017-0460-9
PubMed Abstract | CrossRef Full Text | Google Scholar
ENCODE Project Consortium (2007). Identifizierung und Analyse von funktionellen Elementen in 1% des menschlichen Genoms durch das ENCODE-Pilotprojekt. Nature 447, 799-816. doi: 10.1038/nature05874
CrossRef Full Text | Google Scholar
ENCODE Project Consortium (2012). Eine integrierte Enzyklopädie der DNA-Elemente im menschlichen Genom. Nature 489, 57-74. doi: 10.1038/nature11247
CrossRef Full Text | Google Scholar
Faulkner, G. J., and Carninci, P. (2009). Altruistische Funktionen für egoistische DNA. Cell Cycle 8, 2895-900. doi: 10.4161/cc.8.18.9536
PubMed Abstract | CrossRef Full Text | Google Scholar
Graur, D. (2017). Eine Obergrenze für den funktionalen Anteil des menschlichen Genoms. Genome Biol. Evol. 9, 1880-1885. doi: 10.1093/gbe/evx121
PubMed Abstract | CrossRef Full Text | Google Scholar
Graur, D., Zheng, Y., Price, N., Azevedo, R. B. R., Zufall, R. A., Elhaik, E., et al. (2013). Über die Unsterblichkeit von Fernsehgeräten: „Funktion“ im menschlichen Genom nach dem evolutionsfreien Evangelium von ENCODE. Genome Biol. Evol. 5, 578-590. doi: 10.1093/gbe/evt028
PubMed Abstract | CrossRef Full Text | Google Scholar
Griffiths, P. E. (2009). In welchem Sinne macht „nichts einen Sinn, außer im Lichte der Evolution“? Acta Biotheor. 57:11. doi: 10.1007/s10441-008-9054-9
PubMed Abstract | CrossRef Full Text | Google Scholar
Ingolia, N. T., Brar, G. A., Stern-Ginossar, N., Harris, M. S., Talhouarne, G. J. S., Jackson, S. E., et al. (2014). Ribosome profiling reveals pervasive translation outside of annotated protein-coding genes. Cell Rep. 8, 1365-1379. doi: 10.1016/j.celrep.2014.07.045
PubMed Abstract | CrossRef Full Text | Google Scholar
Ji, Z., Song, R., Regev, A., and Struhl, K. (2015). Many lncRNAs, 5’UTRs, and pseudogenes are translated and some are likely to express functional proteins. eLife 4:e08890. doi: 10.7554/eLife.08890
PubMed Abstract | CrossRef Full Text | Google Scholar
Kim, K. H., Son, J. M., Benayoun, B. A., and Lee, C. (2018). Das mitochondrial-kodierte Peptid MOTS-c transloziert in den Zellkern und reguliert die nukleäre Genexpression als Reaktion auf metabolischen Stress. Cell Metab. 28, 516.e7-524.e7. doi: 10.1016/j.cmet.2018.06.008
PubMed Abstract | CrossRef Full Text | Google Scholar
Knibbe, C., Coulon, A., Mazet, O., Fayard, J. M., and Beslon, G. (2007). Ein langfristiger evolutionärer Druck auf die Menge der nicht-kodierenden DNA. Mol. Biol. Evol. 24, 2344-2353. doi: 10.1093/molbev/msm165
PubMed Abstract | CrossRef Full Text | Google Scholar
Kondo, T., Hashimoto, Y., Kato, K., Inagaki, S., Hayashi, S., and Kageyama, Y. (2007). Kleine Peptid-Regulatoren der Aktin-basierten Zellmorphogenese, kodiert von einer polycistronischen mRNA. Nat. Cell Biol. 9, 660-665. doi: 10.1038/ncb1595
PubMed Abstract | CrossRef Full Text | Google Scholar
Lauressergues, D., Couzigou, J. M., Clemente, H. S., Martinez, Y., Dunand, C., Bécard, G., et al. (2015). Primäre Transkripte von microRNAs kodieren regulatorische Peptide. Nature 520, 90-93. doi: 10.1038/nature14346
PubMed Abstract | CrossRef Full Text | Google Scholar
Li, H., Xiao, L., Zhang, L., Wu, J., Wei, B., Sun, N., et al. (2018). FSPP: A tool for genome-wide prediction of smORF-encoded peptides and their functions. Front. Genet. 9:96. doi: 10.3389/fgene.2018.00096
PubMed Abstract | CrossRef Full Text | Google Scholar
Lu, S., Zhang, J., Lian, X., Sun, L., Meng, K., Chen, Y., et al. (2019). Ein verstecktes menschliches Proteom, kodiert von „nicht-kodierenden“ Genen. Nucleic Acids Res. 47, 8111-8125. doi: 10.1093/nar/gkz646
PubMed Abstract | CrossRef Full Text | Google Scholar
Lynch, M., Bobay, L. M., Catania, F., Gout, J. F., and Rho, M. (2011). The repatterning of eukaryotic genomes by random genetic drift. Annu. Rev. Genomics Hum. Genet. 12, 347-366. doi: 10.1146/annurev-genom-082410-101412
PubMed Abstract | CrossRef Full Text | Google Scholar
Mackowiak, S. D., Zauber, H., Bielow, C., Thiel, D., Kutz, K., Calviello, L., et al. (2015). Umfassende Identifizierung und Analyse von konservierten kleinen ORFs in Tieren. Genome Biol. 16:179. doi: 10.1186/s13059-015-0742-x
PubMed Abstract | CrossRef Full Text | Google Scholar
Magny, E. G., Pueyo, J. I., Pearl, F. M. G., Cespedes, M. A., Niven, J. E., Bishop, S. A., et al. (2013). Konservierte Regulation der kardialen Kalziumaufnahme durch Peptide, die in kleinen offenen Leserahmen kodiert sind. Science 341, 1116-1120. doi: 10.1126/science.1238802
PubMed Abstract | CrossRef Full Text | Google Scholar
Nelson, B. R., Makarewich, C. A., Anderson, D. M., Winders, B. R., Troupes, C. D., Wu, F., et al. (2016). Ein Peptid, das von einem als lange nicht-kodierende RNA annotierten Transkript kodiert wird, erhöht die SERCA-Aktivität im Muskel. Science 351, 271-275. doi: 10.1126/science.aad4076
PubMed Abstract | CrossRef Full Text | Google Scholar
Ohta, T. (2002). Beinahe-Neutralität in der Evolution von Genen und Genregulation. Proc. Natl. Acad. Sci. U.S.A. 99, 16134-16137. doi: 10.1073/pnas.252626899
PubMed Abstract | CrossRef Full Text | Google Scholar
Olexiouk, V., Crappé, J., Verbruggen, S., Verhegen, K., Martens, L., and Menschaert, G. (2016). sORFs.org: a repository of small ORFs identified by ribosome profiling. Nucleic Acids Res. 44, D324-D329. doi: 10.1093/nar/gkv1175
PubMed Abstract | CrossRef Full Text | Google Scholar
Pang, Y., Liu, Z., Han, H., Wang, B., Li, W., Mao, C., et al. (2020). Das Peptid SMIM30 fördert die Entwicklung von HCC durch Induktion der SRC/YES1-Membranverankerung und Aktivierung des MAPK-Wegs. J. Hepatol. doi: 10.1016/j.jhep.2020.05.028. .
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
Polycarpou-Schwarz, M., Groß, M., Mestdagh, P., Schott, J., Grund, S. E., Hildenbrand, C., et al. (2018). Das krebsassoziierte Mikroprotein CASIMO1 kontrolliert die Zellproliferation und interagiert mit der Squalen-Epoxidase und moduliert die Bildung von Lipidtröpfchen. Oncogene 37, 4750-4768. doi: 10.1038/s41388-018-0281-5
PubMed Abstract | CrossRef Full Text | Google Scholar
Pueyo, J. I., and Couso, J. P. (2008). Die 11-Aminosäuren langen Tarsen-losen Peptide lösen ein Zellsignal in der Beinentwicklung von Drosophila aus. Dev. Biol. 324, 192-201. doi: 10.1016/j.ydbio.2008.08.025
PubMed Abstract | CrossRef Full Text | Google Scholar
Pueyo, J. I., and Couso, J. P. (2011). Tarsal-lose Peptide kontrollieren die Notch-Signalisierung durch den Shavenbaby-Transkriptionsfaktor. Dev. Biol. 355, 183-1936. doi: 10.1016/j.ydbio.2011.03.033
PubMed Abstract | CrossRef Full Text | Google Scholar
Quinn, J. J., and Chang, H. Y. (2016). Einzigartige Merkmale der Biogenese und Funktion langer nicht-kodierender RNAs. Nat. Rev. Genet. 17, 47-62. doi: 10.1038/nrg.2015.10
PubMed Abstract | CrossRef Full Text | Google Scholar
Ray, S., Rosenberg, M. I., Chanut-Delalande, H., Decaras, A., Schwertner, B., Toubiana, W., et al. (2019). The mlpt/Ubr3/Svb module comprises an ancient developmental switch for embryonic patterning. eLife 8:e39748. doi: 10.7554/eLife.39748
PubMed Abstract | CrossRef Full Text | Google Scholar
Ridley, M. (2004). Evolution, 3rd edn. Oxford: Blackwell Pub.
Google Scholar
Ruiz-Orera, J., Verdaguer-Grau, P., Villanueva-Cañas, J. L., Messeguer, X., and Alb,à, M. M. (2018). Translation von neutral evolvierenden Peptiden bietet eine Grundlage für de novo Gen-Evolution. Nat. Ecol. Evol. 2, 890-896. doi: 10.1038/s41559-018-0506-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Savard, J., Marques-Souza, H., Aranda, M., and Tautz, D. (2006). Ein Segmentierungsgen in Tribolium produziert eine polycistronische mRNA, die für mehrere konservierte Peptide kodiert. Cell 126, 559-569. doi: 10.1016/j.cell.2006.05.053
PubMed Abstract | CrossRef Full Text | Google Scholar
Singh, U., and Wurtele, E. S. (2020). How new genes are born. eLife 2020:e55136. doi: 10.7554/eLife.55136
PubMed Abstract | CrossRef Full Text | Google Scholar
Tautz, D., and Domazet-Lošo, T. (2011). Der evolutionäre Ursprung von Orphan-Genen. Nat. Rev. Genet. 12, 692-702. doi: 10.1038/nrg3053
PubMed Abstract | CrossRef Full Text | Google Scholar
Tobias-Santos, V., Guerra-Almeida, D., Mury, F., Ribeiro, L., Berni, M., Araujo, H., et al. (2019). Multiple Rollen des polycistronischen Gens Tarsal-Less/Mille-Pattes/Polished-Rice während der Embryogenese der Küssenden Wanze Rhodnius prolixus. Front. Ecol. Evol. 7:379. doi: 10.3389/fevo.2019.00379
CrossRef Full Text | Google Scholar
Van De Peer, Y., Mizrachi, E., and Marchal, K. (2017). Die evolutionäre Bedeutung der Polyploidie. Nat. Rev. Genet. 18, 411-424. doi: 10.1038/nrg.2017.26
PubMed Abstract | CrossRef Full Text | Google Scholar
Vanderperre, B., Lucier, J. F., Bissonnette, C., Motard, J., Tremblay, G., Vanderperre, S., et al. (2013). Die direkte Detektion von alternativen Open Reading Frames Übersetzungsprodukten beim Menschen erweitert das Proteom signifikant. PLoS ONE 8:e70698. doi: 10.1371/journal.pone.0070698
PubMed Abstract | CrossRef Full Text | Google Scholar
Vassallo, A., Palazzotto, E., Renzone, G., Botta, L., Faddetta, T., Scaloni, A., et al. (2020). Der kleine ORF trpM von Streptomyces coelicolor stimuliert Wachstum und morphologische Entwicklung und übt entgegengesetzte Effekte auf die Actinorhodin- und Calcium-abhängige Antibiotika-Produktion aus. Front. Microbiol. 11:224. doi: 10.3389/fmicb.2020.00224
PubMed Abstract | CrossRef Full Text | Google Scholar
Wu, Q., Wright, M., Gogol, M. M., Bradford, W. D., Zhang, N., and Bazzini, A. (2020). Translation von kleinen Downstream-ORFs verstärkt die Translation von kanonischen offenen Hauptleserahmen. EMBO J. 39:e104763. doi: 10.15252/embj.2020104763
PubMed Abstract | CrossRef Full Text | Google Scholar