Frontiers in Genetics
Introduction
Small Open Reading Frames (kleine ORFs/sORFs/smORFs) zijn belangrijke bronnen van mogelijke peptiden die vroeger werden afgedaan als niet-functioneel of junk DNA, zoals bepaald door vroege genvoorspellingsmethoden. In feite zijn smORFs van <100 codons mogelijke coderende sequenties maar voldoende klein om zeer frequent en willekeurig in genomen voor te komen; de opsporing van hun coderend potentieel en functionele beoordeling is dus vergelijkbaar met een wandeling in het duister. Bovendien, terwijl tientallen smORF peptiden recent beschreven zijn als essentiële spelers in biologische processen, worden vele gerapporteerd als potentiële niet-functionele producten van junk DNA onder alomtegenwoordige translatie, wat leidt tot de vraag: vanuit welk perspectief wordt dit gebrek aan functie beoordeeld? In deze context werd onlangs gesuggereerd dat niet-functionele smORF peptiden een belangrijke rol zouden kunnen spelen tijdens de de novo eiwitcoderende gengeboorte, maar het evolutionaire mechanisme is nog onduidelijk. De rol van alomtegenwoordige translatie van smORFs in moleculaire evolutie blijft dus raadselachtig. Hier presenteren we interessante vragen voor debat en verder onderzoek over het perspectief van niet-functionele smORF peptiden als ondergewaardeerde hotspots van moleculaire evolutie in eukaryoten.
Small Open Reading Frames: A Subtopic in the Discussion of Junk DNA Function
Met betrekking tot de evolutie van moleculaire functie, accumuleert een deel van de DNA-elementen mutaties door genetische drift; dus, de evolutie van deze elementen is niet-adaptief en neutraal (Ohta, 2002). In sommige gevallen is de hoeveelheid neutraal evoluerende elementen in junk-DNA analoog aan de items op een menu dat beschikbaar is voor natuurlijke selectie (Knibbe et al., 2007; Faulkner en Carninci, 2009; Lynch et al., 2011). Interessant is dat het ENCODE-consortium (de Encyclopedia of DNA Elements) heeft gerapporteerd dat het grootste deel van het menselijke junk-DNA een vorm van biochemische activiteit vertoont (ENCODE Project Consortium, 2012), maar geen adaptieve relevantie en selectieve druk uitoefent (Doolittle, 2013; Graur et al., 2013). Belangrijk is dat junk DNA 75-90% van het menselijk genoom vertegenwoordigt (Graur, 2017).
Een deel van het junk DNA menu bestaat uit neutraal evoluerende smORF peptiden. Zo worden duizenden niet-coderende RNA’s gegenereerd door de uitgebreide transcriptiedekking op junk DNA (ENCODE Project Consortium, 2007). Toenemend bewijs toont aan dat duizenden smORFs doordringende translatie ondergaan in transcripten die zijn geannoteerd als niet-coderend of in niet-vertaalde regio’s (UTR) van mRNA’s (bijv., Aspden et al., 2014; Ingolia et al., 2014). Interessant is dat niet-coderende RNA’s en ORF’s zonder homologe werden gerapporteerd als kandidaten voor de novo evolutie van eiwitcoderende genen (Tautz en Domazet-Lošo, 2011). Bovendien werd onlangs gesuggereerd dat neutraal evoluerende smORF-peptiden een belangrijke rol zouden kunnen spelen in dit proces (Ruiz-Orera et al., 2018), maar het evolutionaire mechanisme moet nog worden bepaald (Ruiz-Orera et al., 2018; Singh and Wurtele, 2020). In deze context vormen twee eerder voorgestelde concepten die worden gebruikt om evolutie van moleculaire functies te bespreken de kern van het junk-DNA-debat: “causale rollen” en “geselecteerde effecten” (Doolittle en Brunet, 2017), die hier zullen worden besproken in de context van smORF’s en eiwitcoderende gengeboorte.
De “causale rol” beschrijft de activiteit die door een neutraal evoluerend element bij toeval wordt uitgevoerd. Bijvoorbeeld, een hypothetische genomische sequentie gegenereerd door een willekeurige nucleotide mutatie om te lijken op een TATA box kan worden herkend en gebonden door transcriptiefactoren, maar triggert geen gentranscriptie (Griffiths, 2009; Graur et al., 2013). Met andere woorden, “causale rollen” zijn niet-adaptieve fenotypes, hun ontstaan is willekeurig, en ze hebben de neiging om snel te verdwijnen tijdens de evolutie. Anderzijds beschrijven “geselecteerde effecten” de verwerving van adaptieve fenotypen op basis van natuurlijke selectie (Graur et al., 2013), zoals canonieke TATA-boxen of ORF’s die worden vertaald in belangrijke eiwitten. Met andere woorden, “geselecteerde effecten” zijn functioneel relevant voor cellen.
Belangrijk is dat, terwijl natuurlijke selectie adaptieve evolutie aandrijft (geselecteerde effecten), het algemeen wordt aanvaard dat genetische drift de junk DNA-evolutie aandrijft, evenals de synonieme modificaties in coderende DNA-sequenties (CDS) en mutaties in UTR’s van mRNA’s (Ridley, 2004).
Discussie
Op basis van de eerder genoemde bewijzen en concepten bespreken we hier een mogelijk eukaryotisch mechanisme waarmee neutraal evoluerende smORFs de proteoomevolutie bevorderen en het evolutionaire belang van smORFs.
Vooreerst, een deel van de rollen die uitgevoerd worden door neutraal evoluerende smORF peptiden gaan mogelijk over van “causale rollen” naar “geselecteerde effecten” onder omgevingsdruk, waardoor hun neutrale fenotypes blootgesteld worden aan natuurlijke selectie en de evolutie van nieuwe coderende genen in gang wordt gezet. Wanneer neutrale smORF-peptiden worden geselecteerd, zijn ze dus niet langer neutraal (Ruiz-Orera et al., 2018). Met andere woorden, neutrale smORF-peptiden kunnen speciale voorgerechten zijn op het junk-DNA-menu die beschikbaar zijn voor natuurlijke selectie (Figuur 1A).
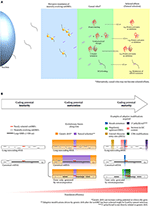
Figuur 1. Fenotype selectie en coderingspotentieel rijping van smORF transcripten. (A) Overgang van smORF-peptiden van “causale rollen” naar “geselecteerde effecten” na pervasieve translatiegebeurtenissen. Pervasieve vertaling van neutraal evoluerende smORFs bevordert mogelijk proteoomevolutie door neutrale fenotypes bloot te stellen aan natuurlijke selectie onder omgevingsdruk. (B) Schema voor coderingspotentiaalrijping, een hypothetisch mechanisme dat de vertalingsefficiëntie van een mRNA verhoogt nadat voor een smORF is geselecteerd (geselecteerd effect) in een transcript met suboptimale coderingskenmerken. Links: onvolwassen coderingspotentieel; in het midden: rijping van het coderingspotentieel; rechts: rijpheid van het coderingspotentieel. Tijdens de coderingspotentieel onvolwassenheidsfase worden nieuw geselecteerde smORFs waargenomen in transcripten met suboptimale coderingskenmerken, hetzij in lange niet-coderende RNAs, hetzij als alternatieve smORFs in canonieke mRNAs. Hoewel canonieke mRNA’s optimale coderingskenmerken vertonen, worden alternatieve smORF’s gewoonlijk secundair of pervasief vertaald; bijgevolg kunnen sommige alternatieve smORF’s zich in suboptimale coderingsgebieden bevinden. Tijdens de rijpingsfase van het coderingspotentieel kunnen natuurlijke selectie en genetische drift in verschillende delen van een transcript optreden. Terwijl natuurlijke selectie de geselecteerde delen vastlegt, verandert genetische drift de niet-coderende delen van een transcript, zoals gepostuleerd door de bijna neutrale theorie (Ohta, 2002). Natuurlijke selectie bevordert fijnafgestemde aanpassingen aan de geselecteerde fenotypes, zoals synonieme mutaties en CDS-modificaties. Genetische drift kan adaptieve mutaties in een transcript tot stand brengen door sequenties te evolueren die de vertaling van smORF’s kunnen verhogen, zoals de Kozak-consensus, regulerende upstream ORF’s, interne ribosoom entry sites (IRES) en verhogingen van het GC-gehalte. Daarnaast kunnen andere adaptieve modificaties die niet direct verband houden met sequentiemutaties in transcripten de expressie van smORF’s verhogen, zoals de 5′ cap, de 3′ poly(A)-staart, cis-regulerende elementen in het genoom en, in het geval van alternatieve smORF’s, de generatie van onafhankelijke geneenheden door retrotranspositie. Belangrijk is dat de verwerving van optimale coderingskenmerken kan worden bevorderd nadat de smORF is geselecteerd, omdat door genetische drift aangedreven modificaties door natuurlijke selectie kunnen worden gefixeerd als zij de translatie-efficiëntie van de nieuw geselecteerde smORF verbeteren. Voordat de smORF geselecteerd is, kunnen eventueel verworven optimale coderingskenmerken snel verdwijnen tijdens de evolutie door genetische drift zonder fixatie. Als alternatief kunnen door genetische drift geëvolueerde mutaties het gen tot zwijgen brengen. Uiteindelijk bereiken smORF’s de rijpheidsfase van het coderingspotentieel wanneer optimale coderingskenmerken verworven zijn en de vertalingsefficiëntie toeneemt. Bijgevolg wordt de vertaalsnelheid van smORF-peptiden na voltooiing van het beschreven proces grotendeels verhoogd, wat bijdraagt aan de totstandkoming van moleculaire innovaties en de geboorte van eiwitcoderende genen.
Op het moment dat smORF’s worden geselecteerd, bevatten ze waarschijnlijk een lage adaptieve relevantie vanwege hun niet-coderende transcriptkenmerken, zoals een lage vertaalsnelheid, gebrek aan 3′-terminale verwerking en andere suboptimale coderingskenmerken (niet-coderende RNA-kenmerken worden besproken in Quinn en Chang, 2016). Deze hypothese is gebaseerd op het feit dat honderden smORF’s worden beschreven als sterk geconserveerd, maar een lage expressie en een lage translatie-efficiëntie vertonen en worden waargenomen in transcripten met niet-coderende kenmerken (Cabili et al., 2011; Aspden et al., 2014; Bazzini et al., 2014). De bijna neutrale theorie (Ohta, 2002) suggereert echter dat niet-coderende delen van vaste smORF-transcripten worden gewijzigd door willekeurige genetische drift, in sommige gevallen kleine voordelige (of nadelige) adaptieve effecten producerend gedurende de evolutie; wij stellen dus voor dat deze modificaties op een bepaald moment het coderingspotentieel van smORF-transcripten verfijnen en verhogen en bijgevolg de adaptieve relevantie van hun peptiden vergroten, zoals te zien is in een groot aantal belangrijke smORF-peptiden die onlangs zijn ontdekt (bijv, Magny et al., 2013; Anderson et al., 2015; Lauressergues et al., 2015; Nelson et al., 2016; Pengpeng et al., 2017; Kim et al., 2018; Polycarpou-Schwarz et al., 2018; Chugunova et al., 2019; Tobias-Santos et al., 2019; Pang et al., 2020; Vassallo et al., 2020). Belangrijk is dat de verwerving van verschillende optimale coderingskenmerken mogelijk wordt bevorderd nadat de smORF is geselecteerd, omdat door genetische drift aangedreven modificaties door natuurlijke selectie kunnen worden gefixeerd als ze de vertaalefficiëntie van de nieuw geselecteerde smORF verbeteren. Voordat de smORF geselecteerd is, kunnen eventueel in de nucleotidesequentie verworven optimale coderingskenmerken snel verdwijnen tijdens de evolutie door genetische drift zonder fixatie. Als alternatief kunnen nucleotideveranderingen het coderingspotentieel negatief beïnvloeden en een gen tot zwijgen brengen. Optimale coderingskenmerken omvatten structurele stabilisatie, het ontstaan van Kozak-consensus, interne ribosoom-entry-sites (IRES), dekking door enhancers en, in sommige gevallen, de verlenging van coderende smORF’s om de CDS (coderende DNA-sequenties) te vergroten (Figuur 1B). Onlangs hebben Couso en Patraquim (2017) voorgesteld dat ten minste een deel van functionele smORFs potentiële de novo precursors van grote CDSs zijn via een stopcodon mutatiepatroon dat “CDS elongatie” wordt genoemd.”
Gezien de veronderstelling dat de werking van evolutie geleidelijk is, stellen wij voor dat het bovengenoemde proces “coding potential maturation” wordt genoemd (figuur 1B). Bijvoorbeeld, smORF translatie wordt op grote schaal gerapporteerd in transcripten met lange niet-coderende RNA (lncRNA) kenmerken (Crappé et al., 2013; Ingolia et al., 2014; Ji et al., 2015; Mackowiak et al., 2015; Li et al., 2018; Lu et al., 2019). Deze lncRNA’s vertonen smORF-conservatie in divergente soorten, wat duidt op fixatie door natuurlijke selectie en duidt op coderende onrijpheid.
Een andere potentiële route van coderende gengeneratie treedt op via alternatieve smORF’s in UTR’s of overlappingen met de referentie-CDS van canonieke mRNA’s. In dit scenario ondergaan alternatieve smORFs alomtegenwoordige translatie of is de translatie zelf belangrijk voor cis-regulerende doeleinden (Vanderperre et al., 2013; Wu et al., 2020). Als de “causale rollen” die worden vervuld door neutraal evoluerende smORF-peptiden “geselecteerde effecten” worden, zouden de alternatieve smORF’s onafhankelijke geneenheden genereren door retrotranspositie, of ze zouden worden gefixeerd als alternatieve smORF’s in de oorspronkelijke transcripten (Figuur 1B). Vandaar dat tijdens retrotranspositiegebeurtenissen, ten minste een deel van de transcripten die op basis van pseudogenisatie zijn onderzocht, in feite de rijping van nieuwe coderende genen kunnen vertegenwoordigen, zoals gesuggereerd door een rapport dat pseudogenen kunnen worden vertaald in zeer geconserveerde smORF-peptiden (Ji et al., 2015).
smORF’s zouden sequentiereservoirs kunnen zijn die mogelijk worden geactiveerd tijdens de evolutie van nieuwe fenotypische variaties, vooral tijdens soortvorming. Belangrijk is dat soortvorming wordt geassocieerd met de evolutie van nieuwe moleculaire fenotypen en nieuwe relaties met de omgeving (Bao et al., 2018). Dus, de hoeveelheid junk DNA en lncRNA’s in cellen verdient onderzoek, niet alleen als een willekeurige opeenhoping van sequenties en translationele ruis, maar ook als een opslagplaats van substraten om de evolutie van nieuwe coderende genen te bevorderen. Interessant is dat polyploïdisatie, of hele genoom duplicatie (WGD) gebeurtenissen, zijn gecorreleerd met een toename van het adaptieve potentieel van cellen en organismen die worden blootgesteld aan stressvolle omstandigheden (Van De Peer et al., 2017). Helaas hebben studies van WGD tot nu toe de rol en het behoud van smORFs tijdens de evolutie verwaarloosd, waarschijnlijk als gevolg van methodologische moeilijkheden bij smORF-identificatie.
Hoewel, de sequencing van verschillende genomen op basis van vergelijkende benaderingen heeft onlangs nieuwe wegen geopend voor smORF-onderzoek. Zo hebben recente evolutionaire studies door onze groep van de smORFs in het mille-pattes/tarsalless/polished rice (mlpt) gen, het meest bekende smORF-bevattende gen bij insecten (Savard et al, 2006; Kondo et al., 2007; Pueyo en Couso, 2008, 2011; Cao et al., 2017; Ray et al., 2019), toonde aan dat een nieuwe ~80 aminozuur smORF (smHemiptera) verscheen tijdens Hemiptera evolutie (Tobias-Santos et al., 2019). Deze smORF in het polycistronische mlpt-mRNA is dus al meer dan 250 miljoen jaar geconserveerd in de groep, en het is niet aanwezig in de genomen van andere insectenordes. We verwachten dat nieuwe vergelijkende analyses van genomen in de toekomst nog meer voorbeelden van orde-specifieke smORFs zullen opleveren, die een ondergewaardeerd reservoir van nieuwe genen en evolutionaire innovaties zouden kunnen vormen.
In het kort, de studie van smORFs is de laatste 5 jaar aanzienlijk toegenomen door recente ontdekkingen van belangrijke smORF peptiden. Dienovereenkomstig heeft de komst van ribosoom profilering de ontdekking mogelijk gemaakt van vele neutraal evoluerende en potentieel niet-functionele smORFs die alomtegenwoordige translatie ondergaan, waarvan de betekenis nog moet worden bepaald (Crappé et al., 2013; Aspden et al., 2014; Bazzini et al., 2014; Olexiouk et al., 2016). In deze context wordt de intrigerende vraag gesteld: waarom zouden cellen energie besteden aan transcriptie en translatie van neutrale en niet-functionele elementen? Er is waarschijnlijk meer dan één antwoord; maar gezien de onderwerpen die in dit artikel worden besproken, stellen we het volgende perspectief voor: wat als de alomtegenwoordige vertaling van neutraal evoluerende smORF-peptiden een elegant mechanisme vormt om proteomevolutie te bevorderen, vooral tijdens speciatiegebeurtenissen? Als dat zo is, dan hebben niet-functionele smORF peptiden een belangrijke functie in evolutionaire zin. Op basis van deze discussie stellen we voor om het concept van functionaliteit te herzien in de context van smORFs.
Bijdragen van auteurs
DG-A en RN hebben in gelijke mate bijgedragen aan het schrijven van dit manuscript. RN heeft bijgedragen aan het verkrijgen van financiering. Alle auteurs droegen bij aan het artikel en keurden de ingediende versie goed.
Funding
RN werd ondersteund door CNPq (307952/2017-7 en 431354/2016-2) en FAPERJ (E-26/210-150/2016, E-26/203.298/2016, E-26/202.605/2019, en E-26/211.169/2019). DG-A was masterstudent van PPG-PRODBIO-UFRJ/Macaé (CAPES-beurs).
Conflict of Interest
De auteurs verklaren dat het onderzoek is uitgevoerd in afwezigheid van commerciële of financiële relaties die als een potentieel belangenconflict zouden kunnen worden opgevat.
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
Bao, R., Dia, S. E., Issa, H. A., Alhusein, D., and Friedrich, M. (2018). Vergelijkend bewijs voor een uitzonderlijke impact van genduplicatie op de ontwikkelingsevolutie van Drosophila en de hogere Diptera. Front. Ecol. Evol. 6:63. doi: 10.3389/fevo.2018.00063
CrossRef Full Text | Google Scholar
Bazzini, A. A., Johnstone, T. G, Christiano, R., Mackowiak, S. D., Obermayer, B., Fleming, E. S., et al. (2014). Identification of small ORFs in vertebrates using ribosome footprinting and evolutionary conservation. EMBO J. 33, 981-993. doi: 10.1002/embj.201488411
PubMed Abstract | CrossRef Full Text | Google Scholar
Cabili, M., Trapnell, C., Goff, L., Koziol, M., Tazon-Vega, B., Regev, A., et al. (2011). Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses. Genes Dev. 25, 1915-1927. doi: 10.1101/gad.17446611
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
Chugunova, A., Loseva, E., Mazin, P., Mitina, A., Navalayeu, T., Bilan, D., et al. (2019). LINC00116 codeert voor een mitochondriaal peptide dat ademhaling en lipidenmetabolisme verbindt. Proc. Natl. Acad. Sci. U.S.A. 116, 4940-4945. doi: 10.1073/pnas.1809105116
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
Doolittle, W. F. (2013). Is junk DNA bunk? Een kritiek op ENCODE. Proc. Natl. Acad. Sci. U.S.A. 110, 5294-5300. doi: 10.1073/pnas.1221376110
CrossRef Full Text | Google Scholar
Doolittle, W. F., and Brunet, T. D. P. (2017). On causal roles and selected effects: our genome is mostly junk. BMC Biol. 15:116. doi: 10.1186/s12915-017-0460-9
PubMed Abstract | CrossRef Full Text | Google Scholar
ENCODE Project Consortium (2007). Identificatie en analyse van functionele elementen in 1% van het menselijk genoom door het ENCODE pilot project. Nature 447, 799-816. doi: 10.1038/nature05874
CrossRef Full Text | Google Scholar
ENCODE Project Consortium (2012). Een geïntegreerde encyclopedie van DNA-elementen in het menselijk genoom. Nature 489, 57-74. doi: 10.1038/nature11247
CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
Graur, D., Zheng, Y., Price, N., Azevedo, R. B. R., Zufall, R. A., Elhaik, E., et al. (2013). Over de onsterfelijkheid van televisietoestellen: “Functie” in het menselijk genoom volgens het evolutievrije evangelie van ENCODE. Genome Biol. Evol. 5, 578-590. doi: 10.1093/gbe/evt028
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
Kim, K. H., Son, J. M., Benayoun, B. A., and Lee, C. (2018). Het mitochondriaal-gecodeerde peptide MOTS-c transloceert naar de kern om nucleaire genexpressie te reguleren in reactie op metabole stress. Cell Metab. 28, 516.e7-524.e7. doi: 10.1016/j.cmet.2018.06.008
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
Kondo, T., Hashimoto, Y., Kato, K., Inagaki, S., Hayashi, S., and Kageyama, Y. (2007). Small peptide regulators of actin-based cell morphogenesis encoded by a polycistronic mRNA. Nat. Cell Biol. 9, 660-665. doi: 10.1038/ncb1595
PubMed Abstract | CrossRef Full Text | Google Scholar
Lauressergues, D., Couzigou, J. M., Clemente, H. S., Martinez, Y., Dunand, C., Bécard, G., et al. (2015). Primaire transcripten van microRNAs coderen regulerende peptiden. Nature 520, 90-93. doi: 10.1038/nature14346
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | Ref Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
Mackowiak, S. D., Zauber, H., Bielow, C., Thiel, D., Kutz, K., Calviello, L., et al. (2015). Uitgebreide identificatie en analyse van geconserveerde kleine ORFs in dieren. Genome Biol. 16:179. doi: 10.1186/s13059-015-0742-x
PubMed Abstract | CrossRef Full Text | Google Scholar
Magny, E. G., Pueyo, J. I., Pearl, F. M. G., Cespedes, M. A., Niven, J. E., Bishop, S. A., et al. (2013). Conserved regulation of cardiac calcium uptake by peptides encoded in small open reading frames. Science 341, 1116-1120. doi: 10.1126/science.1238802
PubMed Abstract | CrossRef Full Text | Google Scholar
Nelson, B. R., Makarewich, C. A., Anderson, D. M., Winders, B. R., Troupes, C. D., Wu, F., et al. (2016). Een peptide gecodeerd door een transcript geannoteerd als lang niet-coderend RNA verhoogt de SERCA-activiteit in spieren. Science 351, 271-275. doi: 10.1126/science.aad4076
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
Olexiouk, V., Crappé, J., Verbruggen, S., Verhegen, K., Martens, L., and Menschaert, G. (2016). sORFs.org: a repository of small ORFs identified by ribosome profiling. Nucleic Acids Res. 44, D324-D329. doi: 10.1093/nar/gkv1175
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
Pengpeng, B., Ramirez-Martinez, A., Li, H., Cannavino, J., McAnally, J. R., Shelton, J. M., et al. (2017). Controle van spiervorming door het fusogene micropeptide myomixer. Science 356, 323-327. doi: 10.1126/science.aam9361
PubMed Abstract | CrossRef Full Text | Google Scholar
Polycarpou-Schwarz, M., Groß, M., Mestdagh, P., Schott, J., Grund, S. E., Hildenbrand, C., et al. (2018). Het kanker-geassocieerde microproteïne CASIMO1 controleert celproliferatie en interageert met squaleen epoxidase modulerend lipide druppelvorming. Oncogene 37, 4750-4768. doi: 10.1038/s41388-018-0281-5
PubMed Abstract | CrossRef Full Text | Google Scholar
Pueyo, J. I., and Couso, J. P. (2008). De 11-aminoacid lange tarsale-loze peptiden triggeren een celsignaal in de pootontwikkeling van Drosophila. Dev. Biol. 324, 192-201. doi: 10.1016/j.ydbio.2008.08.025
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
CrossRef Full Text | Google Scholar
Van De Peer, Y., Mizrachi, E., and Marchal, K. (2017). De evolutionaire betekenis van polyploïdie. Nat. Rev. Genet. 18, 411-424. doi: 10.1038/nrg.2017.26
PubMed Abstract | CrossRef Full Text | Google Scholar
Vanderperre, B., Lucier, J. F., Bissonnette, C., Motard, J., Tremblay, G., Vanderperre, S., et al. (2013). Directe detectie van alternatieve open leesramen translatieproducten in de mens breidt het proteoom aanzienlijk uit. PLoS ONE 8:e70698. doi: 10.1371/journal.pone.0070698
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar