Fronteras de la Genética
Introducción
Los Pequeños Marcos de Lectura Abiertos (small ORFs/sORFs/smORFs) son fuentes importantes de péptidos putativos previamente descartados como ADN no funcional o basura, según determinaron los primeros métodos de predicción de genes. De hecho, los smORFs de <100 codones son posibles secuencias codificantes, pero lo suficientemente pequeñas como para aparecer con mucha frecuencia y de forma aleatoria en los genomas; por tanto, la detección de su potencial codificador y la evaluación funcional es similar a un paseo en la oscuridad. Además, aunque recientemente se han descrito docenas de péptidos smORF como actores esenciales en los procesos biológicos, se ha informado de que muchos de ellos son posibles productos no funcionales del ADN basura sometido a una traducción omnipresente, lo que lleva a preguntarse: ¿desde qué perspectiva se evalúa esta falta de función? En este contexto, se ha sugerido recientemente que los péptidos smORF no funcionales podrían desempeñar un papel importante durante el nacimiento de genes codificantes de proteínas de novo, pero el mecanismo evolutivo aún no está claro. Así pues, el papel de la traducción omnipresente de los smORF en la evolución molecular sigue siendo desconcertante. Aquí, presentamos cuestiones interesantes para el debate y la investigación adicional sobre la perspectiva de los péptidos smORF no funcionales como puntos calientes subestimados de la evolución molecular en eucariotas.
Pequeños marcos de lectura abiertos: Un subtema en la discusión de la función del ADN basura
Respecto a la evolución de la función molecular, parte de los elementos de ADN acumulan mutaciones por deriva genética; así, la evolución de estos elementos es no adaptativa y neutral (Ohta, 2002). En algunos casos, la cantidad de elementos de evolución neutra en el ADN basura son análogos a los elementos de un menú disponible para la selección natural (Knibbe et al., 2007; Faulkner y Carninci, 2009; Lynch et al., 2011). Curiosamente, el consorcio ENCODE (la Enciclopedia de Elementos de ADN) informó de que la mayor parte del ADN basura humano presenta algún tipo de actividad bioquímica (Consorcio del Proyecto ENCODE, 2012), pero carece de relevancia adaptativa y de presión selectiva (Doolittle, 2013; Graur et al., 2013). Es importante destacar que el ADN basura representa el 75-90% del genoma humano (Graur, 2017).
Parte del menú de ADN basura está compuesto por péptidos smORF de evolución neutra. Por ejemplo, miles de ARN no codificantes son generados por la amplia cobertura de transcripción en el ADN basura (Consorcio del Proyecto ENCODE, 2007). Cada vez hay más pruebas de que miles de smORFs se traducen de forma generalizada en los transcritos anotados como no codificantes o en las regiones no traducidas (UTR) de los ARNm (por ejemplo, Aspden et al., 2014; Ingolia et al., 2014). Curiosamente, se ha informado de que los ARN no codificantes y los ORF que carecen de homólogos son candidatos a la evolución de novo de los genes codificantes de proteínas (Tautz y Domazet-Lošo, 2011). Además, recientemente se sugirió que los péptidos smORF de evolución neutra podrían desempeñar un papel importante en este proceso (Ruiz-Orera et al., 2018), pero el mecanismo evolutivo aún está por determinar (Ruiz-Orera et al., 2018; Singh y Wurtele, 2020). En este contexto, dos conceptos propuestos previamente y utilizados para discutir la evolución de las funciones moleculares están en el centro del debate sobre el ADN basura: «roles causales» y «efectos seleccionados» (Doolittle y Brunet, 2017), que se discutirán aquí en el contexto de los smORF y el nacimiento de genes codificantes de proteínas.
El «rol causal» describe la actividad realizada por un elemento que evoluciona neutralmente por azar. Por ejemplo, una secuencia genómica hipotética generada por una mutación aleatoria de nucleótidos para parecerse a una caja TATA puede ser reconocida y unida por factores de transcripción, pero no desencadena la transcripción de genes (Griffiths, 2009; Graur et al., 2013). En otras palabras, los «roles causales» son fenotipos no adaptativos, su aparición es aleatoria y tienden a desaparecer rápidamente durante la evolución. Por otro lado, los «efectos seleccionados» describen la adquisición de fenotipos adaptativos basados en la selección natural (Graur et al., 2013), como las cajas TATA canónicas o los ORF que se traducen en proteínas importantes. En otras palabras, los «efectos seleccionados» son funcionalmente relevantes para las células.
Es importante destacar que, mientras la selección natural impulsa la evolución adaptativa (efectos seleccionados), está ampliamente aceptado que la deriva genética impulsa la evolución del ADN basura, así como las modificaciones sinónimas en las secuencias de ADN codificante (CDS) y las mutaciones en las UTR de los ARNm (Ridley, 2004).
Discusión
Aplicando las evidencias y conceptos mencionados, discutimos aquí un posible mecanismo eucariótico por el cual los smORFs que evolucionan neutralmente avanzan en la evolución del proteoma y la importancia evolutiva de los smORFs.
En primer lugar, parte de las funciones desempeñadas por los péptidos smORF de evolución neutra posiblemente transitan de «funciones causales» a «efectos seleccionados» bajo presión ambiental, exponiendo así sus fenotipos neutros a la selección natural y desencadenando la evolución de nuevos genes codificantes. Así, cuando los péptidos smORF neutros son seleccionados, dejan de ser neutros (Ruiz-Orera et al., 2018). En otras palabras, los péptidos smORF neutros pueden ser entradas especiales en el menú del ADN basura que están disponibles para la selección natural (Figura 1A).
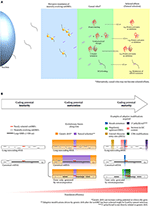
Figura 1. Selección del fenotipo y maduración del potencial de codificación de los transcritos smORF. (A) Transición de los péptidos smORF de «papeles causales» a «efectos seleccionados» tras eventos de traducción generalizada. La traducción omnipresente de los smORF de evolución neutra posiblemente hace avanzar la evolución del proteoma al exponer los fenotipos neutros a la selección natural bajo presión ambiental. (B) Esquema de la maduración del potencial de codificación, un mecanismo hipotético que aumenta la eficiencia de traducción de un ARNm después de que un smORF haya sido seleccionado (efecto seleccionado) en un transcrito con características de codificación subóptimas. A la izquierda, inmadurez del potencial de codificación; en el centro, maduración del potencial de codificación; a la derecha, madurez del potencial de codificación. Durante la fase de inmadurez del potencial de codificación, se observan nuevos smORFs seleccionados en transcritos con características de codificación subóptimas, ya sea en ARN largos no codificantes o como smORFs alternativos en ARNm canónicos. Aunque los ARNm canónicos presentan características de codificación óptimas, los smORFs alternativos suelen traducirse de forma secundaria o generalizada; por tanto, algunos smORFs alternativos pueden residir en regiones de codificación subóptimas. Durante la fase de maduración del potencial de codificación, la selección natural y la deriva genética pueden actuar en diferentes partes de un transcrito. Mientras que la selección natural actúa fijando las partes seleccionadas, la deriva genética actúa cambiando las partes no codificantes de un transcrito, como postula la teoría de la casi neutralidad (Ohta, 2002). La selección natural promueve ajustes finos en los fenotipos seleccionados, como las mutaciones sinónimas y las modificaciones de los CDS. La deriva genética puede establecer mutaciones adaptativas en un transcrito mediante la evolución de secuencias que potencialmente aumentan la traducción del smORF, como el consenso de Kozak, los ORFs reguladores aguas arriba, los sitios de entrada al ribosoma interno (IRES) y el aumento del contenido de GC. Además, otras modificaciones adaptativas no relacionadas directamente con mutaciones en la secuencia de los transcritos podrían aumentar la expresión de los smORF, como la tapa 5′, la cola 3′ de poli(A), los elementos reguladores cis en el genoma y, en el caso de los smORF alternativos, la generación de unidades génicas independientes por retrotransposición. Es importante destacar que la adquisición de características de codificación óptimas podría verse favorecida después de que el smORF haya sido seleccionado, ya que las modificaciones impulsadas por la deriva genética podrían ser fijadas por la selección natural si mejoran la eficiencia de traducción del nuevo smORF seleccionado. Antes de que se haya seleccionado el smORF, las eventuales características de codificación óptimas adquiridas podrían desaparecer rápidamente durante la evolución por deriva genética sin fijación. Alternativamente, las mutaciones evolucionadas por deriva genética pueden silenciar el gen. Finalmente, los smORFs alcanzan la fase de madurez del potencial de codificación cuando se adquieren las características de codificación óptimas y aumenta la eficiencia de traducción. En consecuencia, la tasa de traducción de los péptidos smORF se incrementa en gran medida al completar el proceso descrito, contribuyendo al establecimiento de innovaciones moleculares y al nacimiento de genes codificantes de proteínas.
Al ser seleccionados los smORF, probablemente contengan una baja relevancia adaptativa debido a sus características de transcripción no codificante, como la baja tasa de traducción, la falta de procesamiento 3′-terminal y otras características de codificación subóptimas (las características de ARN no codificante se revisan en Quinn y Chang, 2016). Esta hipótesis se basa en el hecho de que cientos de smORFs se describen como altamente conservados pero muestran una baja expresión, una baja eficiencia de traducción y se observan en transcripciones con características no codificantes (Cabili et al., 2011; Aspden et al., 2014; Bazzini et al., 2014). Sin embargo, la teoría de la casi neutralidad (Ohta, 2002) sugiere que las partes no codificantes de los transcritos smORF fijos se modifican por deriva genética aleatoria, en algunos casos, produciendo pequeños efectos adaptativos ventajosos (o desventajosos) a lo largo de la evolución; así, proponemos que, en un momento dado, estas modificaciones refinan y elevan el potencial codificador de los transcritos smORF y, en consecuencia, aumentan la relevancia adaptativa de sus péptidos, como se observa en un gran número de importantes péptidos smORF descubiertos recientemente (por ejemplo, Magny et al., 2013; Anderson et al., 2015; Lauressergues et al., 2015; Nelson et al., 2016; Pengpeng et al., 2017; Kim et al., 2018; Polycarpou-Schwarz et al., 2018; Chugunova et al., 2019; Tobias-Santos et al., 2019; Pang et al., 2020; Vassallo et al., 2020). Es importante destacar que la adquisición de varias características de codificación óptimas podría verse favorecida después de que el smORF haya sido seleccionado, porque las modificaciones impulsadas por la deriva genética podrían ser fijadas por la selección natural si mejoran la eficiencia de traducción del smORF recién seleccionado. Antes de que el smORF haya sido seleccionado, las eventuales características óptimas de codificación adquiridas en la secuencia de nucleótidos podrían desaparecer rápidamente durante la evolución por deriva genética sin fijación. Alternativamente, los cambios de nucleótidos pueden afectar negativamente al potencial de codificación y silenciar un gen. Las características óptimas de codificación incluyen la estabilización estructural, la aparición de consensos de Kozak, los sitios de entrada al ribosoma interno (IRES), la cobertura de los potenciadores y, en algunos casos, el alargamiento de los smORF codificantes para ampliar las CDS (secuencias de ADN codificantes) (Figura 1B). Recientemente, Couso y Patraquim (2017) propusieron que al menos una parte de los smORFs funcionales son potenciales precursores de novo de grandes CDSs a través de un patrón de mutación de codones de parada llamado «elongación de CDS».»
Considerando la suposición de que la acción de la evolución es gradual, proponemos que el mencionado proceso sea llamado «maduración del potencial de codificación» (Figura 1B). Por ejemplo, se ha informado ampliamente de la traducción de smORF en transcritos con características de ARN no codificante largo (lncRNA) (Crappé et al., 2013; Ingolia et al., 2014; Ji et al., 2015; Mackowiak et al., 2015; Li et al., 2018; Lu et al., 2019). Estos lncRNAs exhiben conservación de smORFs en especies divergentes, lo que insinúa una fijación por selección natural e indica inmadurez de codificación.
Otra vía potencial de generación de genes codificantes ocurre a través de smORFs alternativos en UTRs o superpuestos a la CDS de referencia de mRNAs canónicos. En este escenario, los smORFs alternativos se someten a una traducción omnipresente o el propio acto de traducción es importante para fines de regulación cis (Vanderperre et al., 2013; Wu et al., 2020). Si las «funciones causales» que desempeñan los péptidos smORF de evolución neutra se convierten en «efectos seleccionados», los smORF alternativos generarían unidades génicas independientes por retrotransposición, o se fijarían como smORF alternativos en los transcritos originales (Figura 1B). Por lo tanto, durante los eventos de retrotransposición, al menos una parte de los transcritos investigados sobre la base de la pseudogenización puede, de hecho, representar la maduración de nuevos genes codificantes, como sugiere un informe de que los pseudogenes pueden traducirse en péptidos smORF altamente conservados (Ji et al., 2015).
Los smORFs podrían ser reservas de secuencias potencialmente activadas durante la evolución de nuevas variaciones fenotípicas, especialmente durante la especiación. Es importante destacar que los eventos de especiación se han asociado con la evolución de nuevos fenotipos moleculares y nuevas relaciones con el medio ambiente (Bao et al., 2018). Por lo tanto, la cantidad de ADN basura y lncRNAs en las células merece ser investigada no solo como una acumulación aleatoria de secuencias y ruido traslacional, sino también como un depósito de sustratos para avanzar en la evolución de nuevos genes codificantes. Curiosamente, la poliploidización, o los eventos de duplicación del genoma completo (WGD), se han correlacionado con un aumento del potencial adaptativo de las células y los organismos expuestos a condiciones estresantes (Van De Peer et al., 2017). Desafortunadamente, hasta ahora, los estudios de WGD han descuidado el papel y la retención de los smORF durante la evolución, probablemente debido a las dificultades metodológicas en la identificación de los smORF.
Sin embargo, la secuenciación de varios genomas basada en enfoques comparativos ha abierto recientemente nuevas vías para la investigación de los smORF. Por ejemplo, los recientes estudios evolutivos realizados por nuestro grupo sobre los smORF en el gen mille-pattes/tarsalless/polished rice (mlpt), el gen que contiene smORF más conocido en insectos (Savard et al, 2006; Kondo et al., 2007; Pueyo y Couso, 2008, 2011; Cao et al., 2017; Ray et al., 2019), mostraron que un nuevo smORF de ~80 aminoácidos (smHemiptera) apareció durante la evolución de los Hemípteros (Tobias-Santos et al., 2019). Así, este smORF en el ARNm mlpt policistrónico se ha conservado durante más de 250 millones de años en el grupo, y no está presente en los genomas de otros órdenes de insectos. Esperamos que nuevos análisis comparativos de genomas en el futuro arrojen ejemplos adicionales de smORFs específicos de cada orden, que podrían constituir una reserva infravalorada de nuevos genes e innovaciones evolutivas.
En resumen, el estudio de los smORFs ha aumentado considerablemente durante los últimos 5 años debido a los recientes descubrimientos de importantes péptidos smORF. En consecuencia, la llegada de los perfiles ribosómicos ha permitido el descubrimiento de muchos smORFs de evolución neutra y potencialmente no funcionales que se traducen de forma generalizada, cuya importancia aún está por determinar (Crappé et al., 2013; Aspden et al., 2014; Bazzini et al., 2014; Olexiouk et al., 2016). En este contexto, se plantea la intrigante pregunta: ¿por qué las células gastarían energía en la transcripción y traducción de elementos neutros y no funcionales? Probablemente haya más de una respuesta; sin embargo, teniendo en cuenta los temas tratados en este artículo, proponemos la siguiente perspectiva: ¿y si la traducción omnipresente de péptidos smORF de evolución neutra compone un elegante mecanismo para avanzar en la evolución del proteoma, especialmente durante los eventos de especiación? Si es así, entonces los péptidos smORF no funcionales muestran una función importante en un sentido evolutivo. Basándonos en esta discusión, sugerimos que se revise el concepto de funcionalidad en el contexto de los smORFs.
Contribuciones de los autores
DG-A y RN contribuyeron por igual a la redacción de este manuscrito. RN contribuyó a la adquisición de fondos. Todos los autores contribuyeron al artículo y aprobaron la versión presentada.
Financiación
RN recibió el apoyo del CNPq (307952/2017-7 y 431354/2016-2) y de la FAPERJ (E-26/210-150/2016, E-26/203.298/2016, E-26/202.605/2019 y E-26/211.169/2019). DG-A fue estudiante de máster del PPG-PRODBIO-UFRJ/Macaé (beca CAPES).
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un potencial conflicto de intereses.
PubMed Abstract | CrossRef Full Text | Google Scholar
Aspden, J. L., Eyre-Walker, Y. C., Phillips, R. J., Amin, U., Mumtaz, M. A. S., Brocard, M., et al. (2014). Extensión de la traducción de pequeños marcos de lectura abiertos revelada por poly-ribo-seq. eLife 3:e03528. doi: 10.7554/eLife.03528
Resumen de PubMed | Texto completo de Cracker | Google Scholar
Bao, R., Dia, S. E., Issa, H. A., Alhusein, D., y Friedrich, M. (2018). Evidencia comparativa de un impacto excepcional de la duplicación de genes en la evolución del desarrollo de Drosophila y los Dípteros superiores. Front. Ecol. Evol. 6:63. doi: 10.3389/fevo.2018.00063
CrossRef Full Text | Google Scholar
Bazzini, A. A., Johnstone, T. G, Christiano, R., Mackowiak, S. D., Obermayer, B., Fleming, E. S., et al. (2014). Identificación de pequeños ORFs en vertebrados utilizando la huella del ribosoma y la conservación evolutiva. EMBO J. 33, 981-993. doi: 10.1002/embj.201488411
PubMed Abstract | CrossRef Full Text | Google Scholar
Cabili, M., Trapnell, C., Goff, L., Koziol, M., Tazon-Vega, B., Regev, A., et al. (2011). Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses. Genes Dev. 25, 1915-1927. doi: 10.1101/gad.17446611
PubMed Abstract | CrossRef Full Text | Google Scholar
Cao, G., Gong, Y., Hu, X., Zhu, M., Liang, Z. C., Huang, L., et al. (2017). Identificación de péptidos sin tarso del gusano de seda Bombyx mori. Appl. Microbiol. Biotechnol. 102, 1809-1822. doi: 10.1007/s00253-017-8708-4
PubMed Abstract | CrossRef Full Text | Google Scholar
Chugunova, A., Loseva, E., Mazin, P., Mitina, A., Navalayeu, T., Bilan, D., et al. (2019). LINC00116 codifica un péptido mitocondrial que vincula la respiración y el metabolismo de los lípidos. Proc. Natl. Acad. Sci. U.S.A. 116, 4940-4945. doi: 10.1073/pnas.1809105116
PubMed Abstract | CrossRef Full Text | Google Scholar
PubMed Abstract | CrossRef Full Text | Google Scholar
Crappé, J., Criekinge, W. V., Trooskens, G., Hayakawa, E., Luyten, W., Baggerman, G., et al. (2013). Combinando la predicción in silico y el perfil de ribosomas en una búsqueda de todo el genoma de nuevos sORFs putativamente codificantes. BMC Genomics 14:648. doi: 10.1186/1471-2164-14-648
PubMed Abstract | CrossRef Full Text | Google Scholar
Doolittle, W. F. (2013). Es el ADN basura una chorrada? Una crítica de ENCODE. Proc. Natl. Acad. Sci. U.S.A. 110, 5294-5300. doi: 10.1073/pnas.1221376110
CrossRef Full Text | Google Scholar
Doolittle, W. F., y Brunet, T. D. P. (2017). Sobre los roles causales y los efectos seleccionados: nuestro genoma es mayormente basura. BMC Biol. 15:116. doi: 10.1186/s12915-017-0460-9
PubMed Abstract | CrossRef Full Text | Google Scholar
El Consorcio del Proyecto ENCODE (2007). Identificación y análisis de elementos funcionales en el 1% del genoma humano por el proyecto piloto ENCODE. Nature 447, 799-816. doi: 10.1038/nature05874
CrossRef Full Text | Google Scholar
El Consorcio del Proyecto ENCODE (2012). Una enciclopedia integrada de elementos de ADN en el genoma humano. Nature 489, 57-74. doi: 10.1038/nature11247
CrossRef Full Text | Google Scholar
Faulkner, G. J., and Carninci, P. (2009). Funciones altruistas para el ADN egoísta. Cell Cycle 8, 2895-900. doi: 10.4161/cc.8.18.9536
PubMed Abstract | CrossRef Full Text | Google Scholar
Graur, D. (2017). Un límite superior en la fracción funcional del genoma humano. Genome Biol. Evol. 9, 1880-1885. doi: 10.1093/gbe/evx121
PubMed Abstract | CrossRef Full Text | Google Scholar
Graur, D., Zheng, Y., Price, N., Azevedo, R. B. R., Zufall, R. A., Elhaik, E., et al. (2013). Sobre la inmortalidad de los televisores: «Función» en el genoma humano según el evangelio libre de evolución de ENCODE. Genome Biol. Evol. 5, 578-590. doi: 10.1093/gbe/evt028
PubMed Abstract | CrossRef Full Text | Google Scholar
Griffiths, P. E. (2009). En qué sentido «nada tiene sentido sino a la luz de la evolución»? Acta Biotheor. 57:11. doi: 10.1007/s10441-008-9054-9
PubMed Abstract | CrossRef Full Text | Google Scholar
Ingolia, N. T., Brar, G. A., Stern-Ginossar, N., Harris, M. S., Talhouarne, G. J. S., Jackson, S. E., et al. (2014). Ribosome profiling reveals pervasive translation outside of annotated protein-coding genes. Cell Rep. 8, 1365-1379. doi: 10.1016/j.celrep.2014.07.045
PubMed Abstract | CrossRef Full Text | Google Scholar
Ji, Z., Song, R., Regev, A., and Struhl, K. (2015). Muchos lncRNAs, 5’UTRs, y pseudogenes se traducen y es probable que algunos expresen proteínas funcionales. eLife 4:e08890. doi: 10.7554/eLife.08890
Abstracto de PubMed | Texto Completo de Cros. | Google Scholar
Kim, K. H., Son, J. M., Benayoun, B. A., y Lee, C. (2018). El péptido codificado por la mitocondria MOTS-c se transloca al núcleo para regular la expresión de genes nucleares en respuesta al estrés metabólico. Cell Metab. 28, 516.e7-524.e7. doi: 10.1016/j.cmet.2018.06.008
PubMed Abstract | CrossRef Full Text | Google Scholar
Knibbe, C., Coulon, A., Mazet, O., Fayard, J. M., y Beslon, G. (2007). Una presión evolutiva a largo plazo sobre la cantidad de ADN no codificante. Mol. Biol. Evol. 24, 2344-2353. doi: 10.1093/molbev/msm165
PubMed Abstract | CrossRef Full Text | Google Scholar
Kondo, T., Hashimoto, Y., Kato, K., Inagaki, S., Hayashi, S., y Kageyama, Y. (2007). Pequeños péptidos reguladores de la morfogénesis celular basada en la actina codificados por un ARNm policistrónico. Nat. Cell Biol. 9, 660-665. doi: 10.1038/ncb1595
PubMed Abstract | CrossRef Full Text | Google Scholar
Lauressergues, D., Couzigou, J. M., Clemente, H. S., Martinez, Y., Dunand, C., Bécard, G., et al. (2015). Los transcritos primarios de los microARNs codifican péptidos reguladores. Nature 520, 90-93. doi: 10.1038/nature14346
PubMed Abstract | CrossRef Full Text | Google Scholar
Li, H., Xiao, L., Zhang, L., Wu, J., Wei, B., Sun, N., et al. (2018). FSPP: Una herramienta para la predicción en todo el genoma de péptidos codificados por smORF y sus funciones. Front. Genet. 9:96. doi: 10.3389/fgene.2018.00096
Resumen de PubMed | CrossRef Full Text | Google Scholar
Lu, S., Zhang, J., Lian, X., Sun, L., Meng, K., Chen, Y., et al. (2019). Un proteoma humano oculto codificado por genes ‘no codificantes’. Nucleic Acids Res. 47, 8111-8125. doi: 10.1093/nar/gkz646
PubMed Abstract | CrossRef Full Text | Google Scholar
Lynch, M., Bobay, L. M., Catania, F., Gout, J. F., y Rho, M. (2011). The repatterning of eukaryotic genomes by random genetic drift. Annu. Rev. Genomics Hum. Genet. 12, 347-366. doi: 10.1146/annurev-genom-082410-101412
PubMed Abstract | CrossRef Full Text | Google Scholar
Mackowiak, S. D., Zauber, H., Bielow, C., Thiel, D., Kutz, K., Calviello, L., et al. (2015). Identificación y análisis extensivo de ORFs pequeños conservados en animales. Genome Biol. 16:179. doi: 10.1186/s13059-015-0742-x
PubMed Abstract | CrossRef Full Text | Google Scholar
Magny, E. G., Pueyo, J. I., Pearl, F. M. G., Cespedes, M. A., Niven, J. E., Bishop, S. A., et al. (2013). Conserved regulation of cardiac calcium uptake by peptides encoded in small open reading frames. Science 341, 1116-1120. doi: 10.1126/science.1238802
PubMed Abstract | CrossRef Full Text | Google Scholar
Nelson, B. R., Makarewich, C. A., Anderson, D. M., Winders, B. R., Troupes, C. D., Wu, F., et al. (2016). A peptide encoded by a transcript annotated as long noncoding RNA enhances SERCA activity in muscle. Science 351, 271-275. doi: 10.1126/science.aad4076
PubMed Abstract | CrossRef Full Text | Google Scholar
Ohta, T. (2002). Near-neutrality in evolution of genes and gene regulation. Proc. Natl. Acad. Sci. U.S.A. 99, 16134-16137. doi: 10.1073/pnas.252626899
PubMed Abstract | CrossRef Full Text | Google Scholar
Olexiouk, V., Crappé, J., Verbruggen, S., Verhegen, K., Martens, L., y Menschaert, G. (2016). sORFs.org: un repositorio de pequeños ORFs identificados por perfiles de ribosomas. Nucleic Acids Res. 44, D324-D329. doi: 10.1093/nar/gkv1175
PubMed Abstract | CrossRef Full Text | Google Scholar
Pang, Y., Liu, Z., Han, H., Wang, B., Li, W., Mao, C., et al. (2020). Peptide SMIM30 promotes HCC development by inducing SRC/YES1 membrane anchoring and MAPK pathway activation. J. Hepatol. doi: 10.1016/j.jhep.2020.05.028. .
PubMed Abstract | CrossRef Full Text | Google Scholar
Pengpeng, B., Ramírez-Martínez, A., Li, H., Cannavino, J., McAnally, J. R., Shelton, J. M., et al. (2017). Control de la formación muscular por el micropéptido fusogénico miomixer. Science 356, 323-327. doi: 10.1126/science.aam9361
PubMed Abstract | CrossRef Full Text | Google Scholar
Polycarpou-Schwarz, M., Groß, M., Mestdagh, P., Schott, J., Grund, S. E., Hildenbrand, C., et al. (2018). La microproteína asociada al cáncer CASIMO1 controla la proliferación celular e interactúa con la escualeno epoxidasa modulando la formación de gotas lipídicas. Oncogene 37, 4750-4768. doi: 10.1038/s41388-018-0281-5
PubMed Abstract | CrossRef Full Text | Google Scholar
Pueyo, J. I., y Couso, J. P. (2008). Los péptidos largos de 11 aminoácidos sin tarso desencadenan una señal celular en el desarrollo de la pata de Drosophila. Dev. Biol. 324, 192-201. doi: 10.1016/j.ydbio.2008.08.025
PubMed Abstract | CrossRef Full Text | Google Scholar
Pueyo, J. I., and Couso, J. P. (2011). Los péptidos sin tarso controlan la señalización Notch a través del factor de transcripción shavenbaby. Dev. Biol. 355, 183-1936. doi: 10.1016/j.ydbio.2011.03.033
PubMed Abstract | CrossRef Full Text | Google Scholar
Quinn, J. J., and Chang, H. Y. (2016). Características únicas de la biogénesis y la función del ARN no codificante largo. Nat. Rev. Genet. 17, 47-62. doi: 10.1038/nrg.2015.10
PubMed Abstract | CrossRef Full Text | Google Scholar
Ray, S., Rosenberg, M. I., Chanut-Delalande, H., Decaras, A., Schwertner, B., Toubiana, W., et al. (2019). El módulo mlpt/Ubr3/Svb comprende un antiguo interruptor de desarrollo para el patrón embrionario. eLife 8:e39748. doi: 10.7554/eLife.39748
Resumen de PubMed | CrossRef Full Text | Google Scholar
Google Scholar
Ruiz-Orera, J., Verdaguer-Grau, P., Villanueva-Cañas, J. L., Messeguer, X., y Alb,à, M. M. (2018). La traducción de péptidos de evolución neutra proporciona una base para la evolución genética de novo. Nat. Ecol. Evol. 2, 890-896. doi: 10.1038/s41559-018-0506-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Savard, J., Marques-Souza, H., Aranda, M., y Tautz, D. (2006). Un gen de segmentación en Tribolium produce un ARNm policistrónico que codifica múltiples péptidos conservados. Cell 126, 559-569. doi: 10.1016/j.cell.2006.05.053
PubMed Abstract | CrossRef Full Text | Google Scholar
Singh, U., and Wurtele, E. S. (2020). How new genes are born. eLife 2020:e55136. doi: 10.7554/eLife.55136
Resumen de PubMed | CrossRef Full Text | Google Scholar
Tautz, D., y Domazet-Lošo, T. (2011). El origen evolutivo de los genes huérfanos. Nat. Rev. Genet. 12, 692-702. doi: 10.1038/nrg3053
PubMed Abstract | CrossRef Full Text | Google Scholar
Tobias-Santos, V., Guerra-Almeida, D., Mury, F., Ribeiro, L., Berni, M., Araujo, H., et al. (2019). Múltiples funciones del gen policistrónico Tarsal-Less/Mille-Pattes/Polished-Rice durante la embriogénesis de la chinche besucona Rhodnius prolixus. Front. Ecol. Evol. 7:379. doi: 10.3389/fevo.2019.00379
CrossRef Full Text | Google Scholar
Van De Peer, Y., Mizrachi, E., y Marchal, K. (2017). El significado evolutivo de la poliploidía. Nat. Rev. Genet. 18, 411-424. doi: 10.1038/nrg.2017.26
Abstracto de PubMed | Texto Completo de la Cruz | Google Scholar
Vanderperre, B., Lucier, J. F., Bissonnette, C., Motard, J., Tremblay, G., Vanderperre, S., et al. (2013). La detección directa de productos de traducción de marcos de lectura abiertos alternativos en humanos amplía significativamente el proteoma. PLoS ONE 8:e70698. doi: 10.1371/journal.pone.0070698
PubMed Abstract | CrossRef Full Text | Google Scholar
Vassallo, A., Palazzotto, E., Renzone, G., Botta, L., Faddetta, T., Scaloni, A., et al. (2020). The Streptomyces coelicolor small ORF trpM stimulates growth and morphological development and exerts opposite effects on actinorhodin and calcium-dependent antibiotic production. Front. Microbiol. 11:224. doi: 10.3389/fmicb.2020.00224
PubMed Abstract | CrossRef Full Text | Google Scholar
Wu, Q., Wright, M., Gogol, M. M., Bradford, W. D., Zhang, N., and Bazzini, A. (2020). La traducción de pequeños ORFs aguas abajo mejora la traducción de los marcos de lectura abierta principales canónicos. EMBO J. 39:e104763. doi: 10.15252/embj.2020104763
PubMed Abstract | CrossRef Full Text | Google Scholar